Forschungsbericht
Im Jahr 2009, mit Beginn der Förderung des SFB 815, war die oxidative Schädigung von zellulären bzw. Organstrukturen etabliert und in Einzelfällen mechanistisch verstanden. Unser neuartiges Konzept verfolgte die Hypothese, die Redox-Regulation als universelles Steuerungselement der Signaltransduktion zu verstehen und es losgelöst vom oxidativen Stress zu betrachten. Wir vermuteten, dass Prinzipien der Redox-Regulation, ähnlich wie das fundamentale Steuerungsprinzip der Phosphorylierung vs. Dephosphorylierung, Zellfunktionen reversibel koordinieren.
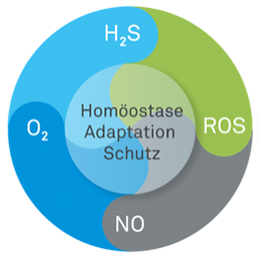
Mit dem integrierenden Prinzip der Redox-Regulation verfolgten wir einerseits innovative Ans√§tze bez√ľglich eines basalen oxidativen Tonus zur Steuerung vitaler Zellfunktionen und andererseits die Idee, dass Redox-Signale zur Zellprotektion (Adaptation, Desensitivierung, Heilung, Schmerz) beitragen, um so √ľberschie√üende pathologische Antworten zu kompensieren. Das Verst√§ndnis der Kommunikation durch Redox-Komponenten bzw. deren funktionelle Konsequenzen sollte zur Identifikation definierter posttranslationaler Proteinmodifikationen von Markerproteinen f√ľhren und mittels Proteomanalysen spezifische Redox-Signaturen aufzeigen. Durch gezielte Ausschaltung, √úberexpression, Hemmung oder selektive Aktivierung von Signalkomponenten im Redox-Signalnetzwerk zielten die Forschungsans√§tze auf eine translationale Komponente ab. Durch die Kombination eines breiten Methodenspektrums, das einen vertikalen Arbeitsansatz vom Enzym/Protein √ľber zellul√§re Systeme bis zum Tierexperiment beinhaltet, war es unser Ziel, auch humanrelevante Fragestellungen zu adressieren. Der Einsatz von Redox-Proteomics zielte darauf ab, neue Komponenten der Redox-Regulation in Form von Zielstrukturen bzw. Signalketten und Proteinclustern zu identifizieren. Der ambitionierte und konzeptionell neue Ansatz, die Redox-Regulation, ausgehend von der Sauerstoffverf√ľgbarkeit und den vielf√§ltigen Reaktionsprodukte, als integratives Steuerungsprinzip physiologischer Signale zu verstehen, war ambitioniert und au√üerordentlich komplex. Unsere Initiative hat Wissenschaftler mit unterschiedlichen Expertisen, Kenntnissen, Forschungsfeldern und methodischen Ans√§tzen von der Grundlagenforschung bis zur Klinik auf der gemeinsamen Interessensplattform der "Redox-Regulation" zusammengef√ľhrt.
Die wachsende Attraktivit√§t, die Redox-Regulation als ein universelles, reversibles Steuerungselement zu verstehen, st√∂√üt oft an technische Limitierungen des Nachweises. Eine einfache Detektion der Redox-Metabolite im biologischen Kontext gestaltet sich schwierig, gezielte Hemmstoffe zeigen oft nicht die gew√ľnschte Selektivit√§t und Redox-Generatoren reflektieren nur unzul√§nglich die Kinetik einer endogenen Redox-Ver√§nderung. Zudem sind die Redox-Metabolite O2-, NO, H2S und O2 in ihren chemischen Reaktionen nicht singul√§r zu betrachten, sondern erzeugen, bedingt durch ihre Reaktivit√§t, eine Vielzahl an Sekund√§rprodukten, die ihrerseits wiederum Redox-Reaktionen eingehen. Diesen Herausforderungen hat sich der SFB 815 gestellt, um Methoden zur Detektion und Quantifizierung von Redox-Ver√§nderungen weiterzuentwickeln, Redox-Knotenpunkte zu identifizieren und deren Mechanismen sowie Funktionen zu entschl√ľsseln.
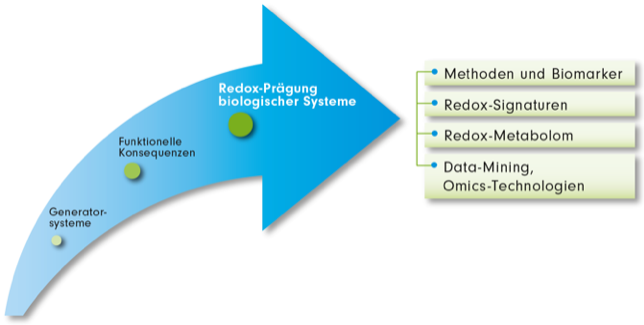
Abb.2W√§hrend in der ersten F√∂rderperiode die Generierung von Redox-Signalen durch NADPH-Oxidasen und Mitochondrien sowie die Rolle von Sauerstoff einschlie√ülich der Hypoxie-vermittelten Genexpression, die Bildung der Sauerstoffmetabolite O2- und NO und deren Interaktion sowie die Produktion von H2S im Vordergrund standen, lag der Arbeitsschwerpunkt der zweiten F√∂rderperiode auf den funktionellen Konsequenzen der Redox-Regulation. Fokus der durch NADPH-Oxidasen und ROS-vermittelten Signale war deren Rolle bei der Zelldifferenzierung sowie Modulation von Schmerz- und Entz√ľndungssignalen. Aspekte der Entz√ľndungsregulation standen auch bei der Redox-Modulation von RNA-Bindeproteinen durch ROS, H2S oder Antioxidantien im Mittelpunkt. Signale, von Sauerstoff/HIF ausgehend, wurden in Bezug auf die Genexpression im Kontext von Infektion und Tumorprogression charakterisiert, w√§hrend ausgew√§hlte Redox-Zielstrukturen bei der Zellkommunikation, Zell√ľberleben, chronischer Entz√ľndung und Schmerz untersucht wurden. In der dritten F√∂rderperiode untersuchten die Teilprojekte die Redox-Pr√§gung biologischer Systeme. Unsere Arbeiten zu den Generatorsystemen hatten uns in die Lage versetzt, ausgew√§hlte Redox-Zielstrukturen molekular zu entschl√ľsseln und distinkte funktionelle Konsequenzen der Redox-modifizierten Zielstrukturen aufzuzeigen. Die Arbeiten zielten darauf ab, zu verstehen, wie ausgew√§hlte Redox-regulierte Zielstrukturen komplexe biologische System pr√§gen. Spezifische Ans√§tze adressierten Probleme, wie Redox-Signale sich auf zellul√§rer Ebene, Organeben und/oder dem Gesamtorganismus sichtbar bzw. detektierbar sind, ob diese Pr√§gungen kausal auf die Redox-Zielstrukturver√§nderungen zur√ľckzuf√ľhren sind und welche Redox-Ph√§notyp√§nderungen als generelle (Bio-)Marker nutzbar sind. Dies beinhaltet Fragen, welche Zellantworten bzw. Krankheits-relevanten Auspr√§gungen urs√§chlich auf reversiblen Redox-Ver√§nderungen basieren. Hierf√ľr ist das Z01-Projekt mit seinen etablierten Methoden, aber auch der Entwicklung neuer Methoden zu Proteomver√§nderungen und Erkennung von Redox-Proteinsignaturen entscheidend beteiligt. Messungen der Redox-Metabolomsignatur sollen helfen, die Pr√§gung biologischer System durch Redox-Signale umfassend zu bearbeiten und zu verstehen.
Der SFB 815 hat darauf hingearbeitet das Zusammenspiel von Generatorsystemen und ausgew√§hlten individuellen funktionellen Konsequenzen zusammenzuf√ľhren, um die Pr√§gung biologischer Systeme durch Redox-Signale zu verstehen. Idealerweise geht es um die Abstraktion individueller Redox-verursachter Funktions√§nderungen f√ľr hierarchisch komplexere Signalabl√§ufe, so dass die Auswirkung singul√§rer Redox-Targetmodifikationen f√ľr Zellen, Organe und ggf. den Gesamtorganismus eingesch√§tzt werden kann. Die von unserem Konsortium vertretene vertikale experimentelle Herangehensweise der Teilprojekte kann nun Aussagen zur Relevanz der Redox-Modifikation f√ľr multifaktoriell regulierte biologische Systeme, z.B. Entz√ľndung, Heilung, Schmerzmodulation, Hypoxie und vaskul√§re Ereignisse liefern. Durch Arbeiten der letzten Jahre sind wir in der Lage die Signifikanz unserer Beobachtungen f√ľr die pathophysiologische Steuerung komplexer Netzwerkstrukturen zuerkennen und in ihrer Relevanz, Allgemeing√ľltigkeit, Robustheit und Tauglichkeit als Marker absch√§tzen. Die Arbeiten legen somit die Basis zum Verst√§ndnis der Pathogenese von Erkrankungen und er√∂ffnen √ľber die spezifische Modulation des Redox-Signalings neue therapeutische Optionen. Mit Hilfe der Erkenntnisse unserer Arbeiten k√∂nnen in der Zukunft Redox-Biomarker etabliert und verifiziert werden und Redox-Signaturen in den in Frankfurt etablierten Forschungsschwerpunkten bestimmt werden. Weiter sollte es m√∂glich sein, vorhandene Daten des Proteoms mit Daten anderer Omics-Technologien zum data mining zu nutzen, um Korrelationen zu metabolischen oder epigenetischen Ver√§nderungen aufzuzeigen.
